Des protéines comme des poissons dans l'eau
©EPFL/iStock photos (luismmolina)
Une méthode informatique développée à l’EPFL permet de prendre explicitement en compte l’impact de l’eau lors de la conception de récepteurs membranaires ayant une stabilité et une signalisation améliorées.
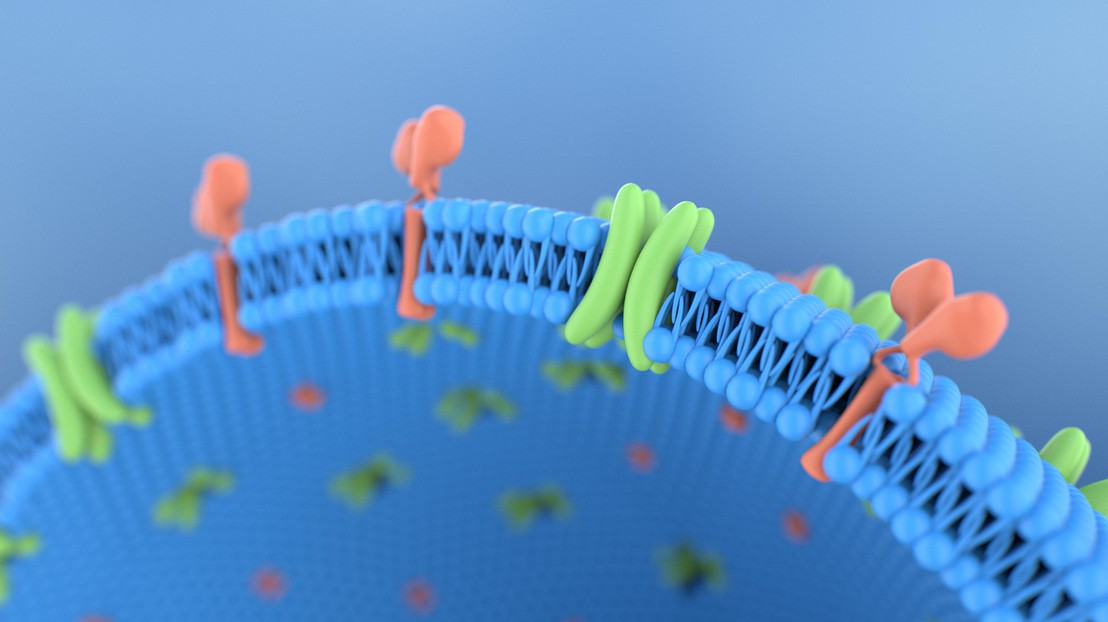
Les protéines sont les moteurs de la vie, alimentant des processus tels que les mouvements musculaires, la vision et les réactions chimiques. Leurs environnements – eau, membranes lipidiques ou autres phases condensées – sont essentiels à leur fonctionnement, façonnant leur structure et leurs interactions.
Pourtant, de nombreuses méthodes modernes de conception des protéines, y compris les outils basés sur l’IA, font souvent l’impasse sur l’influence de ces environnements sur les protéines. Ce qui limite notre capacité à créer des protéines dotées de nouvelles fonctions, freinant les progrès possibles.
Les récepteurs membranaires constituent un groupe de protéines particulièrement sensibles à leur environnement spécifique. Telles des «antennes» biologiques, ils détectent les signaux de l’environnement et en déclenchant des réponses cellulaires.
Les récepteurs couplés aux protéines G (RCPG), précisément, jouent un rôle central dans la perception des cellules des stimuli externes et la manière dont elles y répondent. Pour effectuer leur signalisation, les RCPG comptent sur une interaction délicate entre la stabilité structurelle, la flexibilité et la liaison de ligands, un équilibrage souvent arbitré par l’eau. Ces interactions permettent aux RCPG de changer de forme et de communiquer les signaux qu’ils reçoivent à la cellule.
L’importance de ces gardiens moléculaires pour le fonctionnement cellulaire est telle qu’environ un tiers des médicaments sur le marché les cible. L’ingénierie des protéines concentre également sur eux ses efforts afin de modifier ces récepteurs pour améliorer l’efficacité des médicaments, développer de nouveaux traitements, voire les réutiliser comme biocapteurs en biologie synthétique.
Le problème? Les RCPG sont incroyablement complexes, et leur subtile dépendance à l’eau pour fonctionner les rend impossibles à fabriquer de manière rationnelle... jusqu’à présent.
Une équipe dirigée par Patrick Barth, professeur au Laboratoire d’ingénierie des protéines et des cellules de l’EPFL, a développé des outils informatiques capables de modifier l’échelle des interactions diffusées par l’eau des RCPG afin de concevoir de nouveaux récepteurs membranaires plus performants que leurs homologues naturels. Publiés dans Nature Chemistry, ces travaux ouvrent la voie à des médicaments plus performants et à de nouveaux outils en biologie synthétique.
«L’eau est omniprésente», explique Lucas Rudden, principal coauteur de l’étude. «C’est l’héroïne méconnue de la fonction protéique, et elle est souvent négligée lors de la conception, en particulier lorsqu’il s’agit de l’allostérie des récepteurs membranaires, car elle est difficile à modéliser explicitement. Nous voulions développer une méthode capable de concevoir de nouvelles séquences tout en considérant l’impact de l’eau dans ces réseaux complexes de liaisons hydrogène, qui sont essentiels pour la médiation des signaux dans la cellule.»

Au cœur de cet effort se trouve un outil de conception informatique appelé SPaDES. Les scientifiques l’ont utilisé pour créer des RCPG synthétiques. En commençant par le récepteur de l’adénosine A2A comme modèle, ils se sont penchés sur la modification de ses «centres de communication», c’est-à-dire les sites d’interactions clés entre les molécules d’eau et les acides aminés. Ces centres agissent comme des tableaux de distribution, relayant l’information à travers la protéine. En concevant des réseaux qui optimisent les connexions par l’eau, l’équipe a créé 14 variantes de récepteurs.
Le logiciel SPaDES leur a permis de simuler la façon dont ces changements affecteraient les formes et les fonctions des récepteurs dans différents états critiques. Après un criblage informatique, l’équipe a ensuite synthétisé les récepteurs les plus prometteurs et testé leurs activités dans les cellules.
Le résultat est remarquable: la densité des interactions diffusées par l’eau s’est révélée être un déterminant clé de l’activité des récepteurs. Ceux ayant le plus grand nombre de ces interactions ont montré une plus grande stabilité et une plus grande efficacité de signalisation. Le design le plus prometteur, appelé Hyd_high7, a même adopté une forme inattendue et imprévue, validant les modèles de conception.
Les 14 nouveaux récepteurs ont dépassé leurs homologues naturels à plusieurs égards, notamment par leur capacité à demeurer stables à des températures élevées et leur capacité accrue à lier les molécules de signalisation. Ces qualités les rendent non seulement fonctionnellement supérieurs, mais aussi plus robustes pour la découverte de médicaments et la biologie synthétique.
Ces travaux recèlent un grand potentiel pour la médecine et la biotechnologie. En permettant la conception précise de récepteurs membranaires, la nouvelle méthode pourrait aboutir à des thérapies mieux ciblées pour des maladies, telles que le cancer et les troubles neurologiques. Au-delà de la médecine, ces récepteurs synthétiques pourraient être utilisés dans des biocapteurs ou d’autres outils de détection des changements environnementaux.
Les résultats remettent également en question les hypothèses classiques sur le fonctionnement des RCPG, révélant une flexibilité inattendue dans leurs réseaux d’interactions diffusés par l’eau. Cela ouvre de nouvelles perspectives pour l’exploration d’un potentiel inexploité de ces protéines, tant dans la nature qu’en laboratoire.
Autres contributeurs
- Baylor College of Medicine
- Lilly Biotechnology Center à San Diego
- Lilly Research Laboratories
National Institutes of Health (NIH)
Eli Lilly and Company
Fonds national suisse de la recherche scientifique (FNS)
Ligue suisse contre le cancer
EPFL
Advanced Photon Source, Département de l’énergie des États-Unis (DOE)
Chen, K.-Y., Lai, J. K., Rudden, L. S. P., Wang, J., Russell, A. M., Conners, K., Rutter, M. E., Condon, B., Tung, F., Kodandapani, L., Chau, B., Zhao, X., Benach, J., Baker, K., Hembre, E. J., Barth, P. (2025). Computational design of highly signaling active membrane receptors through solvent-mediated allosteric networks. Nature Chemistry 23 January 2025. DOI: 10.1038/s41557-024-01719-2