Une plateforme d'IA open source facilite la conception de protéines
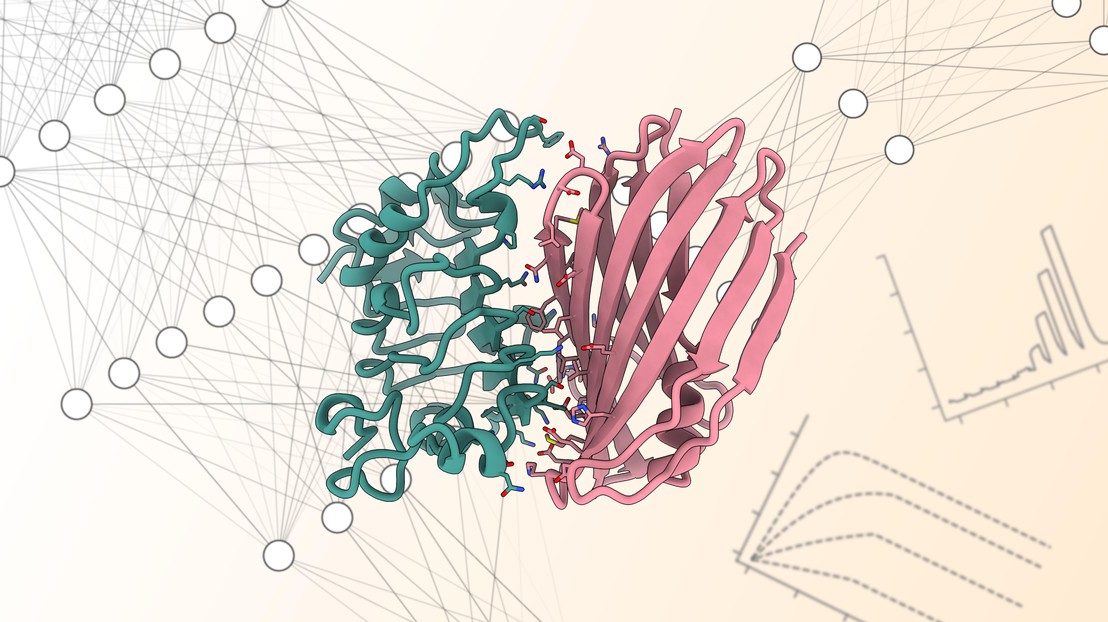
Illustration d'un liant (rose) interagissant avec sa protéine cible (verte). 2025 LPDI EPFL CC BY SA
Depuis sa sortie l’année dernière, BindCraft, la plateforme open source développée à l’EPFL, a déjà bouleversé le monde de la conception de protéines.
Les interactions physiques entre les protéines influencent tout. Que ce soit la signalisation et la croissance cellulaires ou les réponses immunitaires. De ce fait, les biologistes sont très intéressés à pouvoir contrôler ces interactions. Pour développer de nouvelles protéines, appelées liants, les scientifiques utilisent des réseaux neuronaux. Ces liants sont conçus pour se fixer à des cibles pertinentes d'un point de vue thérapeutique, de la même manière que notre système immunitaire se sert d’anticorps pour se lier aux agents pathogènes. Mais ces systèmes, qui utilisent l’apprentissage profond pour prédire la forme des protéines à partir de séquences de blocs constitutifs d’acides aminés, requièrent une expertise en informatique.
«Les méthodes traditionnelles de découverte de liants impliquent l’identification (criblage) de dizaines de milliers de protéines candidates. Ceci nécessite des capacités expérimentales et une expertise informatique que tous les laboratoires ne peuvent pas avoir ou ne possèdent pas», indique Lennart Nickel, doctorant au Laboratoire de conception de protéines et d’immuno-ingénierie (LPDI), dirigé par Bruno Correia, de la Faculté des sciences et techniques de l’ingénieur de l’EPFL. «BindCraft est né de la volonté de développer un outil plus accessible et plus convivial qui n’aurait besoin que de tester une poignée de protéines pour obtenir un liant.»
Avec BindCraft, nous faisons appel à la rétro-ingénierie en utilisant le réseau de prédiction de la structure des protéines dès le départ pour générer de nouveaux liants qui possèdent les propriétés que nous recherchons
Au lieu d’introduire des séquences d’acides aminés dans un réseau neuronal et de cribler minutieusement les liants obtenus pour trouver un résultat satisfaisant, l’équipe de l’EPFL, en collaboration avec des scientifiques du MIT, a utilisé des structures introduites dans le système AlphaFold2 de Google DeepMind. Il permet de générer des séquences pour de nouveaux liants en fonction d’un ensemble de propriétés fonctionnelles souhaitées, comme la liaison à une cible spécifique.
Rétro-ingénierie
«Avec BindCraft, nous faisons appel à la rétro-ingénierie en utilisant le réseau de prédiction de la structure des protéines dès le départ pour générer de nouveaux liants qui possèdent les propriétés que nous recherchons», explique le doctorant Christian Schellhaas.
En se concentrant sur un petit nombre de conceptions de liants, plutôt que sur le criblage à haut débit de vastes bibliothèques de candidats, BindCraft rend la conception de protéines plus efficace et plus accessible. Fruits d’une collaboration entre scientifiques de Suisse, des États-Unis et des Pays-Bas, les résultats ont été publiés dans Nature.
Pour son étude, l’équipe a validé des liants conçus pour interagir avec une douzaine de molécules biotechnologiques et thérapeutiques. Elle a notamment travaillé sur des virus adénoassociés (AAV), utilisés pour livrer des gènes thérapeutiques dans des cellules cibles, la nucléase CRISPR-Cas9, exploitée dans des applications d’édition génomique, ainsi que certains allergènes courants. Dans l’ensemble, les expériences ont montré que les liants de l’équipe se fixaient à leurs cibles avec un taux de réussite moyen de 46 %, offrant la possibilité d’un meilleur contrôle thérapeutique.
Cibler la qualité plutôt que la quantité
«Pour les AAV, l’idée est d’utiliser ces nouveaux liants pour permettre l’administration de gènes uniquement à des cellules et tissus spécifiques, tout en minimisant le risque d’effets secondaires potentiels. Dans le cas de la nucléase CRISPR-Cas9, nos liants peuvent arrêter son activité d’édition génomique et l’empêcher d’agir quand et où elle ne le devrait pas», détaille Martin Pacesa, principal auteur et chercheur au LPDI.
L’automne dernier, une première version de BindCraft sous forme de prépublication, avait rapidement été adoptée par la communauté de biologistes, suscitant des demandes de modifications et de fonctionnalités supplémentaires.
«Nous avons été surpris par la rapidité avec laquelle notre outil a été adopté. Il est même déjà utilisé dans l’industrie. Les demandes des utilisatrices et utilisateurs nous incitent à continuer à développer notre méthode. Nous travaillons déjà à l’adaptation de BindCraft à des molécules d’intérêt thérapeutique plus petites, comme les peptides», précise Martin Pacesa.
Pacesa, M., Nickel, L., Schellhaas, C. et al. One-shot design of functional protein binders with BindCraft. Nature (2025). https://doi.org/10.1038/s41586-025-09429-6