redHUMAN: décoder des liens entre les gènes et le métabolisme
Crédit: Maria Masid (EPFL)
Des scientifiques de l’EPFL ont conçu une méthode qui simplifie le traitement de données génétiques et métaboliques en détectant des modifications du métabolisme indicatives de nombreuses maladies, cancers et Alzheimer notamment. Appelée redHUMAN, il s’agit d’une méthode fiable, offrant une prédictibilité garantie.
Au cours des deux dernières décennies, les partenariats entre les sciences du vivant et les technologies de l’information se sont multipliés. Cette évolution est principalement motivée par le besoin de traiter et d’intégrer de considérables volumes de données provenant de différents domaines parmi lesquels la génétique, la biochimie, la biologie cellulaire et moléculaire, ainsi que la physiologie, afin d’acquérir une meilleure compréhension de systèmes et de processus biologiques, voire d’organismes entiers.
Le problème est qu’il s’est avéré trop compliqué de collecter des données issues de nombreux réseaux biologiques interconnectés à travers différentes strates d’analyse biologique (par exemple génétique ou biochimique). Le volume, mais aussi la complexité des données interdisciplinaires étant difficile à standardiser et à traiter, c’est une des raisons de la prolifération de différents domaines qualifiés d’« -omiques » (génomique, transcriptomique, protéomique, métabolomique, etc.) qui cherchent à caractériser et à quantifier des groupes de molécules selon leur structure et leur fonction dans un organisme.
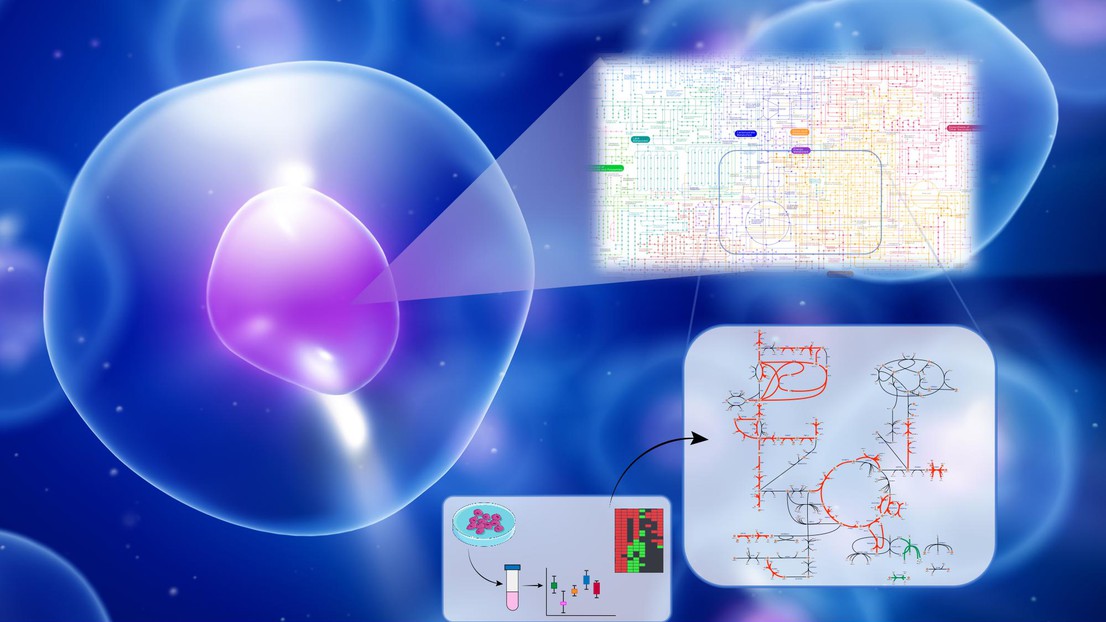
L’une des façons dont les scientifiques ont abordé ce problème dans le contexte de la génétique et de l’analyse métabolique a été de concevoir des modèles de prédiction métabolique à l’échelle du génome (les GEM). Il s’agit là de modèles informatiques établis sur la base de données génétiques et biochimiques, associant les gènes et les voies métaboliques de la cellule.
Les GEM sont de plus en plus couramment utilisés par les chercheurs. « Il s’agit d’outils puissants, très utiles pour intégrer les données expérimentales d’une physiologie spécifique et construire des modèles contextuels capables d’identifier des changements dans le métabolisme de cellules pathologiques comme les cellules cancéreuses », explique la doctorante Maria Masid, du laboratoire de Vassily Hatzimanikatis à l’EPFL.
Maria Masid et ses collègues, qui cherchent à simplifier davantage les GEM, viennent de publier dans la revue Nature Communications un article où ils présentent une nouvelle méthode mathématique permettant d’analyser le métabolisme humain en réduisant la complexité des GEM humains, mettant simplement pour cela l’accent sur certaines parties du métabolisme tout en réduisant la perte de données provenant d’autres voies.
L’étude du métabolisme cellulaire est extrêmement précieuse, les altérations métaboliques étant désormais reconnues pour indiquer différentes pathologies humaines parmi lesquelles le cancer, le diabète, l’obésité, la maladie d’Alzheimer et les maladies cardio-vasculaires. C’est pourquoi le fait de comprendre les liens existants entre les gènes et les mécanismes métaboliques pourrait contribuer à la découverte de nouveaux médicaments et à la conception de nouvelles thérapies.
Les chercheurs, qui ont appelé leur méthode redHUMAN, la décrivent comme « un processus permettant de reconstruire des modèles réduits axés sur les parties du métabolisme intéressant une physiologie spécifique. » RedHUMAN génère des modèles métaboliques à taille réduite incluant les voies présentant un intérêt et les voies métaboliques nécessaires pour étudier le métabolisme nutrimentaire et la synthèse de la biomasse, tout en tenant compte de la bioénergie cellulaire. De cette façon, le modèle redHUMAN garantit la cohérence de ses prédictions, résolvant ainsi un des obstacles majeurs rencontrés par les GEM actuels.
« En croisant ces modèles métaboliques avec des données d’expression génique, nous pouvons repérer des changements fonctionnels que les données ne permettent pas de mettre directement en évidence », souligne Maria Masid, ajoutant « nous pouvons également formuler des hypothèses permettant de mettre au point le plan d’expériences. »
EU Horizon 2020 (Marie-Sklodowska Curie, ITN SyMBioSys)
Maria Masid, Meric Ataman, Vassily Hatzimanikatis. Analysis of human metabolism by reducing the complexity of the genome-scale models using redHUMAN. Nature Communications 11, 2821 (2020). DOI: 10.1038/s41467-020-16549-2.