Prédire le comportement des protéines en regardant leur apparence
L'interaction entre les protéines est à la base de toute ce qui est lié au vivant © Laura Persat / 2019 EPFL
Des chercheurs EPFL ont mis au point une méthode qui permet de prédire exactement les interactions d’une protéine avec d’autres protéines ou biomolécules, ainsi que son activité biochimique, simplement en observant sa surface. Leur méthode open source ouvre de nouvelles perspectives pour la création de protéines artificielles.
La façon dont les protéines interagissent avec leur environnement est à la base de tout processus lié au vivant, et au cœur de tout système biologique. Comprendre ces phénomènes est donc capital pour développer des médicaments efficaces, ou poser les fondations pour la conception de cellules artificielles.
Il existe à présent un système basé sur une technique d’apprentissage automatique, qui permet de prédire ces interactions, et de décrire l’activité biochimique des protéines simplement en observant leur surface. Cette méthode inédite a été développée par des chercheurs du Laboratoire de conception de protéines et d’immuno-ingénierie, à
l’Institut de bioingénierie de la Faculté des sciences et techniques de l’ingénieur de l’EPFL, en collaboration avec USI-Lugano, Imperial College et la section Graph Learning Research de Twitter.
Appelée MaSIF, la nouvelle méthode ouvre de nouvelles perspectives pour comprendre le fonctionnement des protéines, et pour concevoir les éléments à base de protéines, pour les cellules artificielles. La recherche est publiée dans Nature Methods.
Une approche basée sur les données
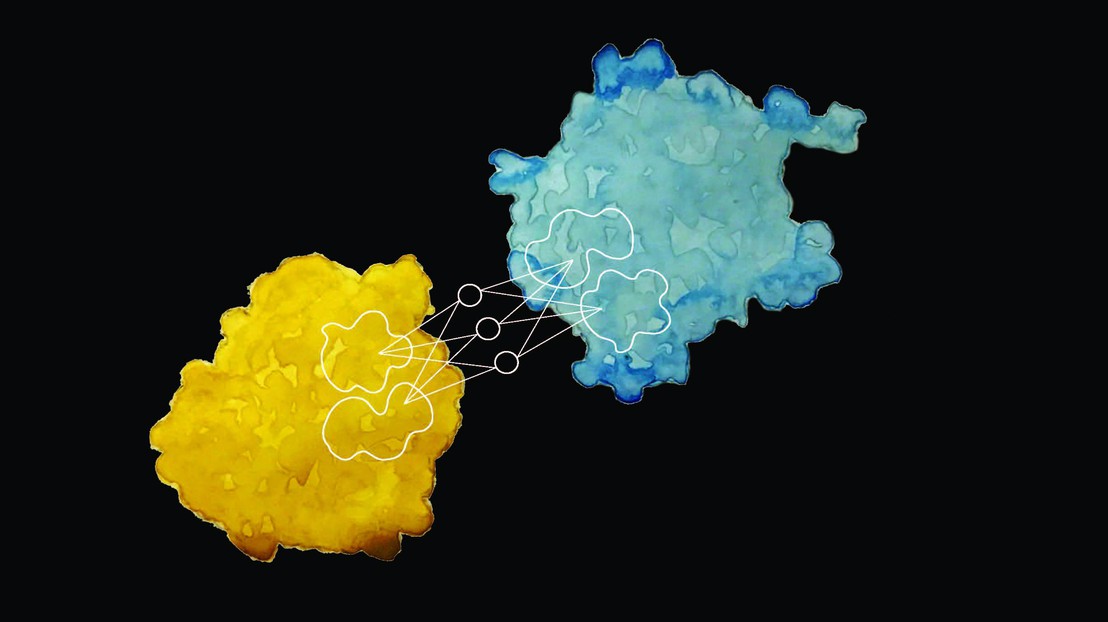
Les chercheurs ont utilisé une large quantité de données relatives à la surface des protéines, et à leurs propriétés géométriques et chimiques. Ils ont entraîné un algorithme de machine-learning à associer ces propriétés à certains comportements et activité biochimique. Les chercheurs ont ensuite vérifié le bon fonctionnement de leur algorithme, avec les données restantes. « En scannant la surface des protéines, notre méthode permet de définir une «empreinte digitale», et de comparer ces empreintes des protéines », indique Pablo Gainza, premier auteur de l’étude.
Les chercheurs ont ainsi constaté que les protéines ayant la même « empreinte » se comportaient de manière similaire.
Crédit: Laura Persat / 2019 EPFL
«Nous pouvons analyser des milliards de surfaces par seconde», explique Bruno Correia, directeur du Laboratoire de conception de protéines et d’immuno-ingénierie. «Pour la conception de protéines artificielles, on pourrait dès lors se référer à cet outil, et programmer le comportement d’une protéine en jouant avec les propriétés géométriques et chimiques de sa surface».
La méthode pourrait par ailleurs être utilisée pour analyser la surface d’autres types de molécules. Les informations sont maintenant à disposition de la communauté scientifique.
P. Gainza, F. Sverrisson, F. Monti, E. Rodolà, D. Boscaini, M. M. Bronstein, and B. E. Correia, “Deciphering interaction fingerprints from protein molecular surfaces using geometric deep learning,” Nature Methods, 2019.