«J'ai eu la chance de tomber sur ce projet et les bonnes personnes»
© Alain Herzog / 2021 EPFL
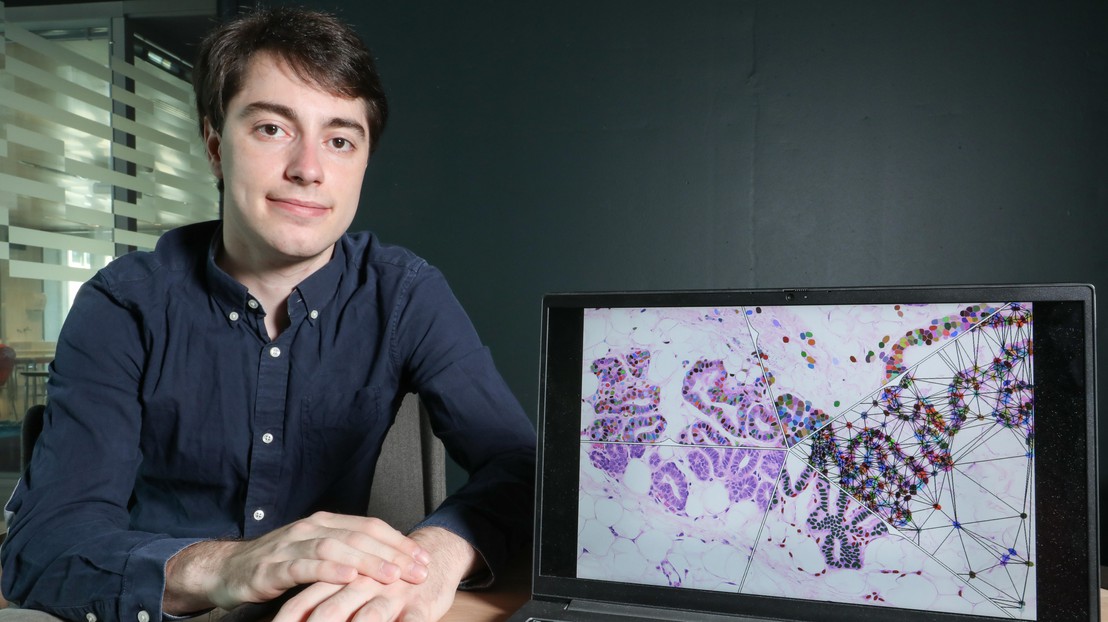
Étudiant en Master d’informatique, Quentin Juppet est premier auteur d’une publication scientifique en biosciences. Avec une équipe transdisciplinaire réunissant biologistes, ingénieurs et informaticiens, il a développé un algorithme permettant de distinguer les cellules humaines de cellules de souris dans une coupe de tissu. Un outil qui facilitera la recherche sur le cancer.
Passionné d’informatique, Quentin Juppet s’est formé très tôt à la programmation. Originaire de Savoie en France, il a suivi ses premiers cours de programmation donnés par des professeurs de l’EPFL alors qu’il n’avait que 15 ans. «J’ai commencé par un cours en ligne pour apprendre à programmer en C++ et l’année d’après, j’ai continué avec un cours sur le langage Java. C’est grâce à ces MOOCs que j’ai connu l’EPFL. Lorsque je suis venu y faire mon bachelor par la suite, j’ai eu la chance de retrouver ces professeurs et je suis même devenu assistant pour l’un d’eux.»
Un outil d’analyse d’images innovant
Aujourd’hui étudiant en 2e année de Master en informatique, il vient de publier son premier article scientifique* réalisé en collaboration avec plusieurs chercheurs de l’EPFL. Transdisciplinaire, la publication est issue d’une collaboration fructueuse entre des biologistes, des ingénieurs et des informaticiens provenant de différentes sections de l’École.
L’article présente un algorithme basé sur le machine learning qui permet de distinguer les cellules humaines de cellules de souris dans une coupe de tissu. Dans le cadre de la recherche sur le cancer, l’étude de cellules cancéreuses peut se faire en greffant des cellules humaines sur des souris. Mais il faut ensuite pouvoir différencier ces deux types de cellules. Habituellement, elle demande l’analyse de multiples tissus avant de trouver les cellules humaines recherchées et de plusieurs colorations par marqueurs fluorescents. Avec l’outil développé par les chercheurs, ce travail est simplifié et la discrimination des cellules se fait automatiquement.
Pour en arriver là, les chercheurs ont d’abord établi une liste d’attributs qui décrivent ces deux types de cellules. «Avec Fabio De Martino, doctorant au Laboratoire de la professeure Cathrin Brisken de la Faculté des sciences de la vie, nous avons retranscrit des données biologiques comme la taille ou la forme en chiffre. En plus de ces descriptions des cellules en elles-mêmes, nous avons pris en compte leur entourage, les cellules contiguës», explique Quentin Juppet. À partir de ces données, l’outil basé sur le machine learning va pouvoir distinguer automatiquement les cellules humaines des cellules de souris. Baptisé Single Cell Classifier (SCC), il est disponible en open source sous forme de plug-in pour le logiciel de traitement et d'analyse d'images ImageJ. Il a également l’avantage de pouvoir être reparamétré pour l'étude d’autres types de cellules.

Un projet qui prend de l’ampleur
À l’origine, Quentin Juppet ne devait effectuer qu’un simple projet étudiant de quatre semaines sur ce thème. «C’était dans le cadre d’un cours de machine learning, donné aux premières années de master de l’EPFL. Avec mon groupe, nous avions travaillé sur ce sujet proposé par le Laboratoire de la professeure Cathrin Brisken, mais n’avions pas eu le temps d’aboutir à quelque chose de directement utile pour la recherche.» Le projet devient alors le projet de semestre de Quentin, mené pendant plusieurs mois.
«J’étais cette fois sous la supervision de Dr. Daniel Sage, senior scientist au Laboratoire d’imagerie biomédicale (STI). Il m’a apporté son expertise pour les algorithmes et l’imagerie en général.» Sous l’égide du tout nouveau EPFL Center for Imaging (ECI), le projet a été mené dans un temps record avec les expertises combinées de Dr. Martin Weigert, group leader (SV) et d’Olivier Burri, image analyst à la plateforme de microscopie (BIOP) où les images à analyser sont réalisées. Un travail qui donnera lieu à la publication mentionnée plus haut. «J’ai eu la chance de tomber sur ce projet et sur les bonnes personnes, d’avoir eu des professeurs pour me guider et l’environnement de l’EPFL qui offre tout à portée de main.»
Lire aussi : Le Centre d'imagerie rassemble les compétences des cinq facultés
Le goût de la recherche
Pour son projet de Master à la Faculté informatique et communications, Quentin Juppet poursuit sur sa lancée et continue à travailler sur l’analyse d’image. «Mon projet se fait au sein de Lunaphore, une spin-off de l'EPFL pour laquelle j’ai travaillé à temps partiel tout au long de mes études. C’était important pour moi de faire de la pratique en parallèle de mes cours.» Bientôt diplômé, Quentin prévoit d'effectuer un doctorat. «Je vais d’abord travailler pendant au moins un an à temps plein pour Lunaphore. Mais par la suite, pourquoi ne pas rester dans l’académique ? J’avoue que cette première publication scientifique m’a donné le goût de la recherche.»
*Juppet, Q., De Martino, F., Marcandalli, E., Weigert, M., Burri, O., Unser, M., Brisken, C., Sage, D. Deep Learning Enables Individual Xenograft Cell Classification in Histological Images by Analysis of Contextual Features. J Mammary Gland Biol Neoplasia (2021). https://doi.org/10.1007/s10911-021-09485-4

