Des analogues de protéines membranaires pour la recherche biomédicale
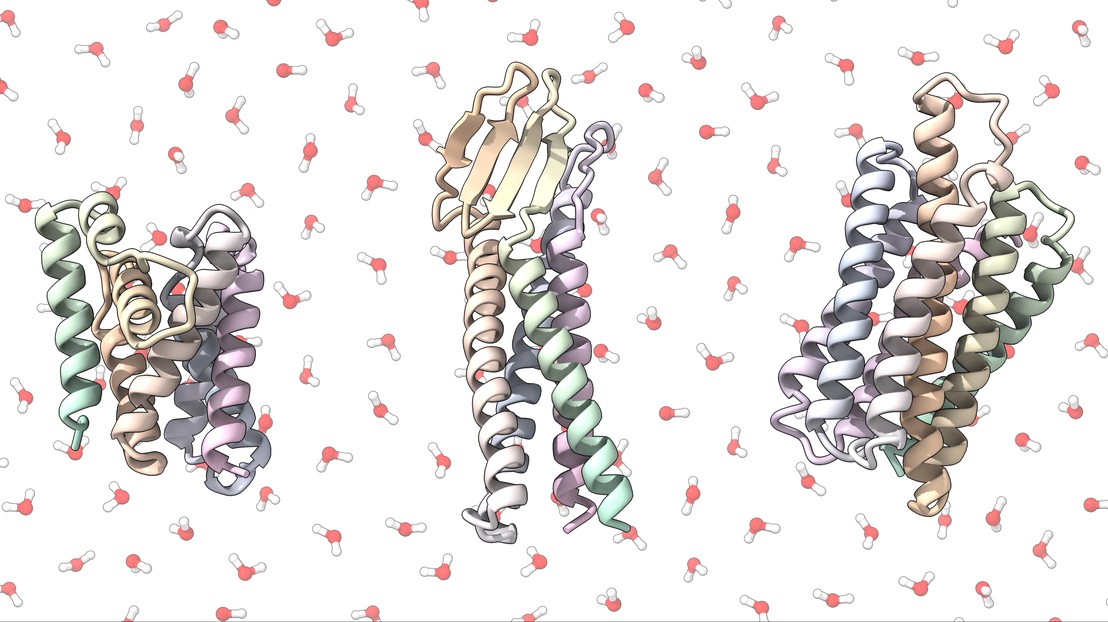
Rendu de certains analogues de protéines solubles de l’équipe. EPFL/LPDI -CC-BY-SA 4.0
Grâce à un pipeline d’apprentissage profond, des scientifiques de l’EPFL ont élaboré des analogues solubles de certaines structures protéiques utilisées dans le développement pharmaceutique. Cette avancée pourrait permettre de contourner le coût prohibitif de l’extraction de ces protéines des membranes cellulaires.
De nombreuses méthodes de découverte de médicaments et d’anticorps ciblent des protéines membranaires au pliage complexe: lorsque les molécules d’un candidat-médicament se lient à ces protéines, elles déclenchent des cascades chimiques qui modifient le comportement cellulaire. Mais comme ces protéines sont intégrées dans la couche externe lipidique des cellules, elles sont difficiles d’accès et insolubles dans les solutions aqueuses (hydrophobes), ce qui les rend difficiles à étudier.
«Nous voulions extraire ces protéines de la membrane cellulaire. Nous les avons donc repensées sous la forme d’analogues solubles hyperstables, qui ressemblent aux protéines membranaires mais sont beaucoup plus faciles à manipuler», explique Casper Goverde, doctorant au Laboratoire de conception de protéines et d’immuno-ingénierie (LPDI) de la Faculté des sciences et techniques de l’ingénieur.
En bref, Casper Goverde et une équipe de recherche du LPDI, sous la houlette de Bruno Correia, ont eu recours à l’apprentissage profond pour élaborer des versions synthétiques solubles de protéines membranaires couramment utilisées dans la recherche pharmaceutique. Alors que les méthodes de criblage traditionnelles reposent sur l’observation indirecte des réactions cellulaires aux candidats-médicaments et candidats-anticorps, ou sur l’extraction minutieuse de petites quantités de protéines membranaires à partir de cellules de mammifères, l’approche computationnelle des scientifiques leur permet de retirer les cellules de l’équation. Après avoir élaboré un analogue de protéine soluble à l’aide de leur pipeline d’apprentissage profond, ils peuvent utiliser des bactéries pour produire en masse la protéine modifiée. Ces protéines peuvent alors se lier directement en solution avec des candidats moléculaires d’intérêt.
«Nous estimons que la production d’un lot d’analogues de protéines solubles avec E. coli est environ 10 fois moins coûteuse qu’avec des cellules de mammifères», ajoute le doctorant Nicolas Goldbach.
Les recherches de l’équipe ont récemment été publiées dans la revue Nature.
Inverser le scénario de l’élaboration des protéines
Ces dernières années, les scientifiques ont réussi à exploiter des réseaux d’intelligence artificielle qui utilisent l’apprentissage profond pour élaborer de nouvelles structures protéiques, par exemple en les prédisant sur la base d’une séquence d’unités structurales (acides aminés). Mais pour cette étude, les scientifiques se sont intéressés à des plis protéiques déjà présents dans la nature; ils avaient besoin d’une version plus accessible et soluble de ces protéines.
«Nous avons eu l’idée d’inverser ce pipeline d’apprentissage profond qui prédit la structure protéique: si nous entrons une structure, peut-il nous indiquer la séquence d’acides aminés correspondante?», interroge Casper Goverde.
L’équipe a utilisé la plateforme de prédiction de structure AlphaFold2 de Google DeepMind pour produire des séquences d’acides aminés pour les versions solubles de plusieurs protéines membranaires essentielles, sur la base de leur structure 3D. Ensuite, elle a utilisé un second réseau d’apprentissage profond, ProteinMPNN, pour optimiser ces séquences et obtenir ainsi des protéines fonctionnelles et solubles. Les scientifiques ont découvert que leur approche était remarquablement fiable et précise dans la production de protéines solubles avec le maintien d’une partie de leur fonctionnalité native, même lorsqu’elle était appliquée à des plis très complexes qui avaient jusqu’à présent échappé à d’autres méthodes de conception.
«Le Graal de la biochimie»
L’étude a notamment permis d’élaborer un analogue soluble d’une forme protéique appelée récepteur couplé aux protéines G (RCPG), qui représente environ 40% des protéines membranaires de l’être humain et constitue une cible pharmaceutique majeure.
«Pour la première fois, nous avons montré qu’il était possible de repenser le RCPG sous la forme d’un analogue soluble stable. C’est un problème de longue date dans le domaine de la biochimie, car si l’on parvient à rendre l’analogue soluble, il est possible de rechercher de nouveaux médicaments beaucoup plus rapidement et facilement», déclare Martin Pacesa, scientifique au LPDI.
Les scientifiques considèrent également ces résultats comme une démonstration de faisabilité pour l’application de leur pipeline à la recherche de vaccins, voire de traitements contre le cancer. Par exemple, ils ont élaboré un analogue soluble d’un type de protéine appelé claudine, qui joue un rôle dans la résistance des tumeurs au système immunitaire et à la chimiothérapie. Dans leurs expériences, l’analogue soluble de la claudine a conservé ses propriétés biologiques, ce qui confirme le potentiel du pipeline à produire des cibles intéressantes pour le développement pharmaceutique.
Goverde, C.A., Pacesa, M., Goldbach, N. et al. Computational design of soluble and functional membrane protein analogues. Nature (2024). https://doi.org/10.1038/s41586-024-07601-y