Contrôler les interactions entre protéines grâce à l'IA
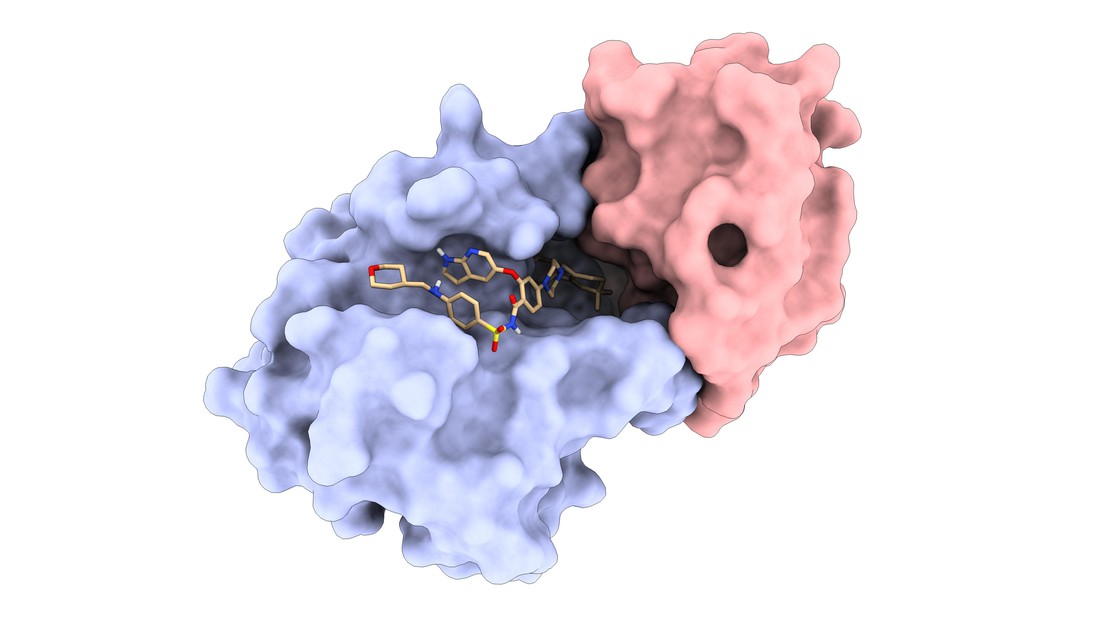
Une des protéines conçues de novo du LPDI (rouge) lié à la protéine Bcl2 (bleu) dans un complexe avec le médicament approuvé par la FDA Vénétoclax (beige) © LPDI EPFL
Des scientifiques de l’EPFL ont utilisé l’IA pour concevoir de nouvelles protéines qui se lient à des complexes impliquant d’autres petites molécules comme des hormones ou des médicaments. Cette avancée ouvre de nombreuses perspectives en matière de conception computationnelle d’interactions moléculaires pour la biomédecine.
En 2023, des scientifiques du Laboratoire de conception de protéines et d’immuno-ingénierie (LPDI) de la Faculté des sciences de l’ingénieur et de la Faculté des sciences de la vie, dirigé par Bruno Correia, ont publié dans la revue Natureun outil basé sur l’intelligence artificielle pour concevoir de nouvelles protéines qui interagissent avec des cibles thérapeutiques. MaSIF peut scanner rapidement des millions de protéines pour identifier les correspondances optimales entre les molécules en fonction de leurs propriétés chimiques et géométriques de surface. Cela permet aux scientifiques de concevoir de nouvelles interactions entre protéines qui jouent un rôle clé dans la régulation cellulaire et les thérapies.
Aujourd’hui, l’équipe a réalisé une autre avancée grâce à cette technologie – également publiée dans la revue Nature. Elle a utilisé MaSIF pour concevoir de nouvelles protéines qui interagissent avec d’autres protéines ayant elles-mêmes une interaction connue avec des petites molécules comme des médicaments ou des hormones. Parce que ces petites molécules induisent des changements subtils dans les propriétés de surface («néosurfaces») des protéines auxquelles elles se lient, elles peuvent agir comme des interrupteurs «ON/OFF» pour le contrôle précis des fonctions cellulaires comme la transcription de l’ADN ou la dégradation des protéines.
«Notre idée était de concevoir une interaction dans laquelle une petite molécule provoque la rencontre de deux protéines. Certaines approches ont visé la découverte de ces petites molécules, mais ici nous voulions concevoir une nouvelle protéine qui se lierait à un complexe protéine-médicament déjà défini», explique Anthony Marchand, scientifique au LPDI et co-premier auteur.
L’équipe a montré que MaSIF pouvait appliquer de manière remarquable des représentations de surface protéique («empreintes digitales»), qui n’avaient été entraînées que sur des protéines, à des néosurfaces émergeant de complexes protéine-médicament. La plupart des systèmes de conception de protéines basés sur l’IA ne fonctionnent que sur des éléments constitués d’acides aminés issus de la nature. Cependant, la sensibilité et la capacité de généralisation de MaSIF aux petites molécules signifient que l’outil pourrait servir à concevoir des interactions entre protéines qui sont induites par stimuli chimiques dans des cellules modifiées; par exemple pour des thérapies cellulaires contrôlées par des médicaments ou des biocapteurs.
Petit mais performant
Bien que l’interaction entre protéines puisse sembler aussi simple que l’assemblage de pièces de puzzle, les variations à la surface des protéines rendent difficile de prédire en réalité où et comment les événements de liaison se produiront. Comme dans leur étude précédente, l’équipe a conçu de nouvelles protéines à l’aide de MaSIF pour générer des «empreintes digitales» pour des caractéristiques de surface telles que la charge positive et négative, l’hydrophobicité ou la forme géométrique. Elle a ensuite identifié des surfaces complémentaires à partir d’une base de données, greffé des fragments protéiques sur des protéines plus grandes et sélectionné des candidats censés être les plus complémentaires à leur cible.
«La différence ici est que nous supposons que les caractéristiques de surface d’une protéine changent si une petite molécule s’y lie, créant ainsi une néosurface. MaSIF a pu saisir cette différence avec une grande sensibilité», indique Arne Schneuing, doctorant au LPDI et co-auteur.
L’équipe a validé expérimentalement ses nouvelles protéines contre trois complexes protéiques liés à des médicaments contenant respectivement l’hormone progestérone, le médicament contre la leucémie approuvé par la FDA Vénétoclax et l’antibiotique naturel actinonine. Les protéines conçues à l’aide de MaSIF ont réussi à reconnaître chaque complexe -protéine-médicament avec une haute affinité. Les scientifiques expliquent que cela a été possible parce que MaSIF est basé sur des caractéristiques de surface générales qui s’appliquent aussi bien aux protéines qu’aux petites molécules. Ils ont donc pu cartographier les caractéristiques des petites molécules dans le même espace descripteur sur lequel MaSIF a été entraîné pour les protéines.
«MaSIF a un nombre relativement faible de paramètres – environ 70 000 contre des milliards pour les grands systèmes d’apprentissage profond comme ChatGPT. Cela est possible parce que nous n’utilisons que des caractéristiques de surface clés, ce qui entraîne un haut niveau d’abstraction. En d’autres termes, nous ne donnons pas au système une image complète mais seulement une partie que nous jugeons importante pour résoudre le problème», précise Arne Schneuing.
Si l’on peut contrôler avec précision l’activité spatio-temporelle des thérapies cellulaires à l’aide d’interrupteurs basés sur des petites molécules, on peut vraiment améliorer l’innocuité et l’efficacité du traitement
Un meilleur contrôle des cellules CAR-T
Une application potentielle intéressante de ces travaux est le contrôle précis des traitements anticancéreux basés sur des cellules comme la thérapie par récepteur antigénique chimérique (CAR-T). Celle-ci consiste à modifier les lymphocytes T d’une patiente ou d’un patient pour mieux cibler son cancer. Mais après avoir été réintroduites chez les patientes et patients, les cellules modifiées peuvent attaquer les mauvaises cibles – provoquant potentiellement des effets secondaires néfastes – ou épuiser leur capacité à lutter contre le cancer. Dans le cadre d’une expérience, l’équipe de l’EPFL a démontré qu’un système inductible au Vénétoclax conçu avec MaSIF était efficace pour activer, à la demande, l’activité de destruction des tumeurs par des cellules CAR-T in vitro.
«Si l’on peut contrôler avec précision l’activité spatio-temporelle des thérapies cellulaires à l’aide d’interrupteurs basés sur des petites molécules, on peut vraiment améliorer l’innocuité et l’efficacité du traitement», résume Stephen Buckley, doctorant au LPDI et co-premier auteur.
Marchand, A., Buckley, S., Schneuing, A. et al. Targeting protein–ligand neosurfaces with a generalizable deep learning tool. Nature (2025). https://www.nature.com/articles/s41586-024-08435-4
Gainza, P., Wehrle, S., Van Hall-Beauvais, A. et al. De novo design of protein interactions with learned surface fingerprints. Nature (2023). https://doi.org/10.1038/s41586-023-05993-x