Améliorer le séquençage de l'ADN en allant moins vite
© 2015 EPFL
Des scientifiques de l’EPFL ont développé une méthode qui améliore la précision du séquençage de l’ADN jusqu’à mille fois. La méthode, qui utilise des nanopores pour lire les nucléotides individuels, ouvre la voie à un séquençage de l’ADN meilleur - et plus économique.
Une lecture trop rapide
L’ADN es une molécule constituée de quatre composants de base qui se répètent. Ils sont appelés nucléotides, et sont assemblés en diverses combinaisons qui contiennent l’information génétique de la cellule, comme les gènes. Fondamentalement, ces quatre nucléotides composent l’ensemble du langage génétique. Le séquençage de l’ADN vise à déchiffrer ce langage, en le réduisant aux lettres individuelles qui le constituent.
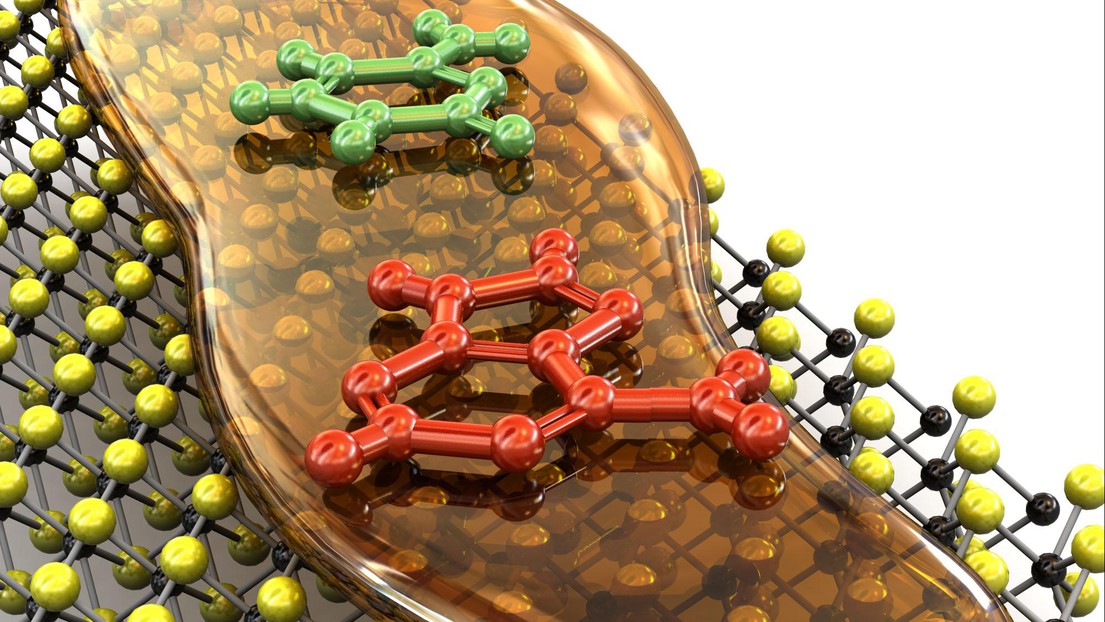
Lors du séquençage par nanopores, l’ADN passe par un minuscule trou dans une membrane, tout comme un fil passe par le chas d’une aiguille. Le pore contient aussi un courant électrique. Lorsque chacun des nucléotides passe au travers du pore, il bloque le courant de manière spécifique, ce qui permet de l’identifier. Bien que puissante, cette méthode souffre de la grande vitesse avec laquelle l’ADN passe à travers les pores, une vitesse trop élevée pour permettre une lecture suffisamment précise.
Ralentir les choses
A l’Institut de Bioengineering de l’EPFL, le laboratoire d’Aleksandra Radenovic a réussi à résoudre le problème lié à la vitesse en utilisant une liquide épais et visqueux, qui ralentit le passage de l’ADN de deux à trois ordres de grandeur. Le résultat est que la précision du séquençage s’abaisse jusqu’au nucléotide individuel.
La recherche a été accomplie par Jiandong Feng et Ke Liu, en collaboration avec des collègues du laboratoire d’Andras Kis à l’EPFL. Les deux chercheurs ont développé un film constitué de disulfure de molybdène (MoS2), d’une épaisseur de seulement 0,7 nm. C’est en soi déjà une innovation par rapport aux expériences qui utilisent du graphène dans ce domaine: l’ADN est une molécule assez collante, tandis que le MoS2 est considérablement moins adhésif que le graphène. L’équipe de chercheurs a créé ensuite un nanopore sur une membrane, large de presque 3 nm.
L’étape suivante consistait à dissoudre l’ADN dans un liquide épais contenant des ions chargés, dont la structure moléculaire peut être ajustée pour moduler son épaisseur, ou son «gradient de viscosité». Le liquide appartient à la classe des «liquides ioniques à température ambiante», qui sont essentiellement des sels dissous dans une solution. Les scientifiques de l’EPFL ont tiré parti de la modularité du liquide pour l’amener à un gradient idéal de viscosité - suffisamment pour ralentir l’ADN.
Pour finir, l’équipe à testé son procédé en faisant passer de multiples fois des nucléotides connus, dissous dans le liquide, au travers du nanopore. Cela leur a permis d’obtenir une mesure moyenne pour chacun des quatre nucléotides, qui peut être utilisée pour les identifier ultérieurement.
Bien qu’ils se trouvent encore dans un processus de tests, les chercheurs de l’équipe prévoient de continuer leur travail en testant des brins entiers d’ADN. «Nous sommes à la recherche de possibilités de commercialisation de cette technique, qui est prometteuse s’agissant de nanopores à l’état solide», dit Jiandong Feng.
Les scientifiques prédisent aussi qu’en recourant à de l’électronique de pointe et à un contrôle très fin du gradient de viscosité, on pourrait encore optimiser le système. En combinant des liquides ioniques avec des nanopores sur des films minces de disulfure de molybdène, ils espèrent aboutirent à une plateforme de séquençage plus économique et offrant de meilleurs résultats.
Ce travail offre un moyen innovant d’améliorer l’une des meilleures méthodes de séquençage existantes. «Dans les années à venir, la technologie deséquençage va clairement migrer de la recherches à la pratique clinique», dit Aleksandra Radenovic. «Pour cela, nous avons besoin d’un séquençage de l’ADN rapide et abordable - ce que la technologie des nanopores peut assurer.»
Ce travail a été effectué entre le Laboratoire de biologie à l'échelle nanométrique et le Laboratoire d'électronique et structures à l'échelle nanométrique de l’EPFL. Il a été financé par le Conseil européen de la recherche.
Source
Feng J, Liu K, Bulushev RD, Khlybov S, Dumcenco D, Kis A, Radenovic A. Identification of single nucleotides in MoS2 nanopores.Nature Nanotechnology 21 September 2015. DOI: 10.1038/nnano.2015.219