Comment les virus infectent les bactéries avec leur dard
© thinkstockphotos
Les bactériophages sont des virus qui infectent les bactéries. Au moyen d’outils ultra-modernes, des scientifiques de l’EPFL ont décrit une «queue» d’un million d’atomes que les bactériophages utilisent pour percer la surface des bactéries. Cette découverte a des implications majeures pour la science et la médecine, puisque les bactériophages sont largement utilisés dans le recherche.
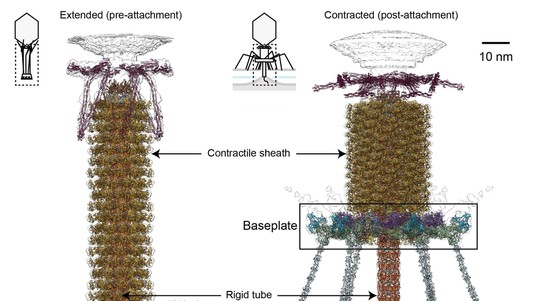
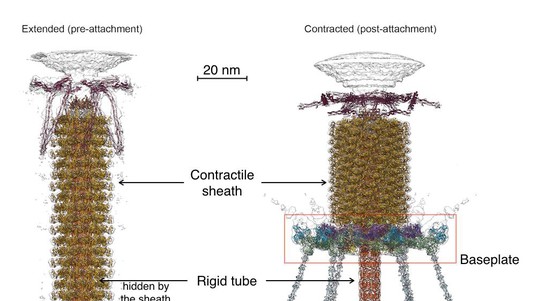
Pour infecter les bactéries, la plupart des bactériophages emploient une «queue» qui frappe et transperce la membrane de la bactérie, afin de permettre au matériel génétique du virus de passer au travers. Les plus sophistiquées de ces pointes consistent en un manchon contractile entourant un tube comparable à un ressort hélicoïdal étiré à l’échelle nanométrique. Lorsque le virus se fixe à la surface de la bactérie, le manchon se contracte et introduit le tube à travers celle-ci. Tout cela est contrôlé par une « base » d’un million d’atomes située au bout de la queue. Des scientifiques de l’EPFL viennent de montrer, à un niveau de détail atomique, comment cette base coordonne la fixation du virus à la bactérie lors de la contraction du manchon de la queue. Cette découverte a fait la couverture de Nature, et elle a d’importantes implications pour la science et la médecine.
Les phages sont largement répandus sur la planète. Ils accompagnent les bactéries partout - dans le sol, dans les eaux, les sources chaudes, dans les proliférations d’algues, dans les intestins des animaux, etc. – et ils ont un impact essentiel sur la diversité des populations bactériennes, y compris par exemple le microbiome de l’intestin humain. Les phages sont également des outils indispensables en génétique et en biologie moléculaire, et ils ont même été développés comme alternative aux antibiotiques. Cependant, les mécanismes par lesquels ces virus s’attachent aux cellules hôtes et délivrent leur matériel génétique demeure mal compris.
Le laboratoire de Petr Leiman à l’EPFL vient de créer un modèle détaillé au niveau atomique de la transformation de la base d’un phage, une structure importante qui contrôle la capacité du virus à trouver sa bactérie-cible et à s’y attacher, contracter sa queue, et injecter son ADN. Le complexe constitué de la base et du tube consiste en un million d’atomes, constituant 145 chaînes de 15 différentes protéines. La plupart d’entre elles ont dû être modélisée à partir de zéro. Pour ce faire, Nicholas Taylor et Ricardo Guerrero-Ferreira, tous deux post-doctorants au laboratoire de Petr Leiman, ont d’abord obtenu des images à haute résolution grâce à l’équipement ultra-moderne du Center for Cellular Imaging and NanoAnalytics (C-CINA) de l’Université de Bâle. Nicholas Taylor, premier auteur de l’article de Nature, les a ensuite analysées au moyen des ressources informatiques du département High Performance Computing de l’EPFL.
Les scientifiques ont également été capables d’identifier un ensemble minimal de composants moléculaires dans la plaque de base qui agissent ensemble comme de minuscules engrenages pour contrôler l’activité de la queue du virus. Ces composants, et le mécanisme fonctionnel sous-jacent, sont les mêmes pour de nombreux virus, et même des bactéries qui utilisent des structures en forme de queue similaires pour injecter des toxines dans les cellules situées à proximité.
«Ces résultats sont importants pour nous permettre de comprendre comment ces systèmes semblables à des queues contractiles fonctionnent,» dit Leiman. «Mais ils marquent aussi une étape dans la complexité des systèmes biologiques qui peuvent être décrit au niveau atomique.» Le corps humain contient presque autant de bactéries que de cellules humaines (30 à 40 trillions), et le microbiote intestinal va vraisemblablement représenter une cible importante pour une médecine personnalisée à l’avenir. «Il est clair que nous avons besoin de comprendre les mécanismes détaillés par lesquels ces bactéries interagissent entre elles, et comment les phages sont impliqués dans ces interactions.»
Cette étude est une collaboration entre leLaboratory of Structural Biology and Biophysics de l’EPFL et leCenter for Cellular Imaging and NanoAnalytics (C-CINA) de l’University de Bâle. Elle a été financée par l’EPFL, l’Université de Bâle, NCCR TransCure, et le Fonds National Suisse.