Des fragments d'ADN révèlent la diversité des espèces en rivières

© 2018 Pixabay
Les traces de matériel génétique présentes dans les rivières permettent de détecter les organismes qui y vivent, sans avoir besoin de les collecter et de les identifier sous le microscope. Des chercheurs de l’Eawag, de l’EPFZ et de l’EPFL ont développé un modèle informatique permettant même, à partir de multiples mesures d’ADN, de simuler la localisation exacte et la fréquence des espèces.
Chaque être vivant laisse des traces infimes de son patrimoine génétique, par exemple sous forme de cellules mortes de la peau ou de déjections. En prélevant des échantillons d’eau et en décryptant l’ADN environnemental (appelé aussi eADN) qu’ils contiennent, on sait quelles espèces fréquentent les eaux correspondantes. Cela permet également de découvrir des espèces rares qui passent littéralement au travers le filet lors d’un échantillonnage normal. Ce concept d’eADN est certes déjà connu depuis un certain temps. Toutefois : « Jusqu’ici, il était possible de déterminer à l’aide de l’eADN la présence ou non d’une espèce. Mais l’on ne connaissait pas la répartition de cette espèce dans l’ensemble de l’écosystème » explique Luca Carraro. Après avoir achevé cet été sa thèse de doctorat en hydrologie de l’environnement à l’EPFL, il effectue désormais des recherches au sein de l’Eawag.
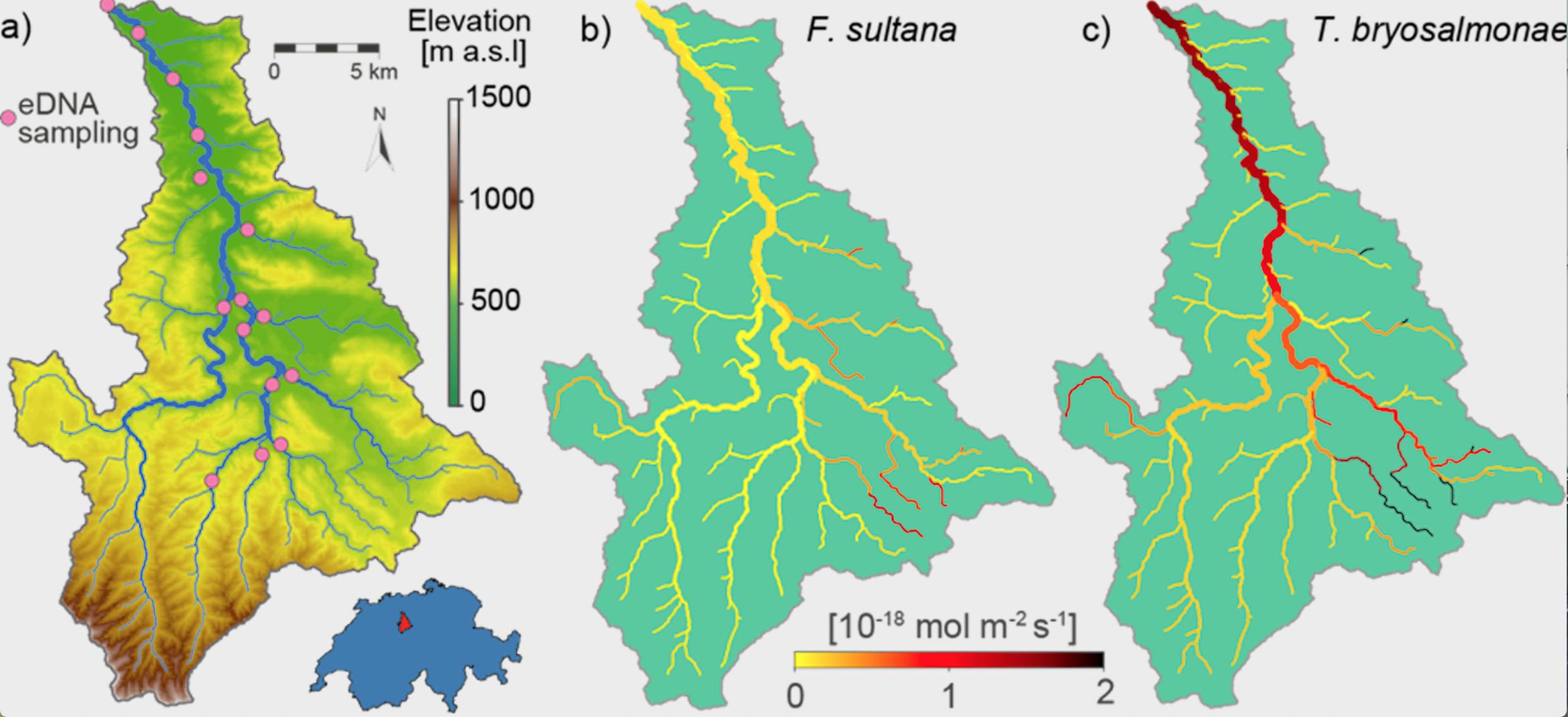
En collaboration avec des chercheurs de l’Eawag, de l’EPFZ et de l’EPFL, il a réussi à reconstituer l’abondante présence d’espèces à partir de mesures ponctuelles d’eADN. Carraro et ses collègues ont pour la première fois utilisé cette méthode pour déterminer la répartition dans la biomasse d’un bryozoaire (Fredericella sultana) et de son parasite (Tetracapsuloides bryosalmonae) dans la Wigger, une rivière suisse préalpine. Ce parasite est le facteur déclenchant de la MRP, une redoutable maladie affectant les poissons, qui provoque en Suisse une hécatombe de truites périssant par milliers.
Le modèle informatique prend en compte l’hydrologie et l’écologie
Afin de pouvoir déterminer avec le plus de précision possible la répartition des organismes, les chercheurs ont développé un modèle informatique basé sur des concepts hydrologiques et écologiques des réseaux hydrographiques. Le modèle est alimenté avec des valeurs de mesures de matériel génétique et prend en compte la dynamique de transport et de dégradation de fragments d’eADN au fil de l’eau ainsi que les facteurs environnementaux. « Cette méthode permettra une surveillance simple et peu coûteuse de la biodiversité dans de vastes régions du monde. Ce qui n’était pas possible jusqu’ici avec les méthodes traditionnelles », Hanna Hartikainen, co-auteure et chef de groupe de recherche à l’Eawag, en est convaincue.
Contact
Stephanie Schnydrig
[email protected]
+41 58 765 6432
Luca Carraro
[email protected]
+41 58 765 5207
Hanna Hartikainen
[email protected]
+41 58 765 5446
Publication originale
Luca Carraroa, Hanna Hartikainenb, Jukka Jokelab, Enrico Bertuzzod, and Andrea Rinaldoa (2018). Estimating species distribution and abundance in river networks using environmental DNA. PNAS, https://doi.org/10.1073/pnas.1813843115
Published online: October 29